Mis à jour le 17 janvier 2021
Médecine personnalisée : pourquoi (et comment) modéliser le cerveau
Élucider les mécanismes qui sous-tendent le fonctionnement de notre cerveau et des maladies qui l’affectent est une étape obligée pour espérer améliorer substantiellement notre santé et notre vie quotidienne.
Dans cette perspective, des décennies de recherche neuroscientifique ont permis d’améliorer les connaissances sur la manière dont notre cerveau accomplit les nombreuses tâches qui lui incombent, de la vision au contrôle des mouvements, en passant par la mémorisation, etc. Cependant, les processus sur lesquels reposent les fonctions cognitives supérieures, telles que l’attention, la prise de décision, voire la conscience, nous échappent encore.
Il en est de même en ce qui concerne notre compréhension des dysfonctionnements du cerveau, qui demeure limitée. Certes, nous comprenons l’impact que peuvent avoir des changements localisés, qu’ils soient d’origine traumatique ou induits par une maladie. Cependant, il nous est encore difficile de saisir les implications plus larges résultant de l’altération des interactions engendrée par ces dommages cérébraux.
Or si nous souhaitons par exemple être capables de mettre au point des traitements efficaces, il est indispensable de mettre au jour les mécanismes qui façonnent et coordonnent l’activité cérébrale globale. Les modèles numériques peuvent nous y aider. Mais créer une réplique numérique d’un organe aussi complexe que le cerveau humain est loin d’être aisé.
Une masse de données à organiser
Grâce aux progrès des neurosciences, nous sommes capables de collecter des quantités croissantes de données, qu’elles proviennent de technologies invasives (sondes, électrodes…) ou non invasives (IRM fonctionnelle, électroencéphalographie, tomographie…), à des résolutions toujours plus fines.
Le défi est aujourd’hui d’organiser et d’intégrer cette formidable masse de données. En nous permettant de formaliser nos connaissances et de les confronter avec nos observations, les modèles numériques du cerveau constituent un outil inestimable pour construire un cadre d’étude cohérent. Ce type d’initiative, intrinsèquement collaborative, est illustrée par le « Human Brain Project » (projet « Cerveau Humain »), dont l’objectif ultime est de bâtir une robuste infrastructure de recherche destinée à faciliter les investigations en neurosciences.
Le projet comporte entre autres le développement d’un « cerveau virtuel » qui pourra être utilisé pour simuler les effets de certaines thérapies, tester les hypothèses concernant le fonctionnement cérébral, etc.
Où en est-on ?
Il existe de nombreuses façons de construire des modèles numériques du cerveau humain. On peut, par exemple, débuter par la base en se focalisant sur le comportement et les interactions de certains neurones spécifiques.
De tels modèles nous permettent de mieux comprendre les principes d’organisation du cerveau, et les fonctions de populations de neurones localisées, ainsi que leurs interconnexions (on parle de « microcircuits neuronaux ») et la façon dont ils traitent les informations qu’ils reçoivent : stimulations sensorielles, comportements moteurs, etc.
Cependant, cette approche ne peut être utilisée pour modéliser le cerveau entier, pour des raisons à la fois techniques et conceptuelles. En particulier, la complexité des connexions neuronales est trop grande (rappelons que le cerveau humain contient de l’ordre de 100 milliards de neurones) : la simulation de régions très restreintes du cerveau nécessite à elle seule de mobiliser les capacités de traitement d’un supercalculateur. Et quand bien même nous disposerions des capacités techniques permettant de recréer numériquement le cerveau entier, neurone par neurone, l’analyse d’un tel système se heurterait aux mêmes défis que l’analyse du cerveau lui-même. Autrement dit, un tel cerveau serait à la simulation cérébrale ce qu’une carte à l’échelle 1 :1 serait à la cartographie : la reproduction la plus détaillée possible, mais la moins pratique qui soit…
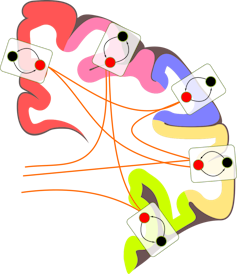
Pour modéliser le cerveau dans son ensemble, une approche complémentaire consiste donc à changer d’échelle, pour se placer au niveau des structures cérébrales. On peut alors envisager le cerveau comme un réseau de diverses populations neuronales dont l’activité individuelle peut être résumée au moyen d’équations.
Pour employer une analogie météorologique, cela revient à décrire l’atmosphère non plus en fonction des molécules qui la composent, mais en termes de températures, pressions et vélocité de l’air. Un tel modèle est calculable à l’échelle du cerveau entier, et fournit une description interprétable des mécanismes de l’activité neuronale. Qui plus est, un tel modèle peut être enrichi par les modèles résultant des données empiriques collectées via l’imagerie cérébrale notamment. Il peut en retour être utilisé pour générer des données simulées, et vérifier si celles-ci sont effectivement en accord avec les observations.
Vers une simulation personnalisée
La personnalisation des simulations est l’un des aspects les plus excitants de ce type de recherche. Il est en effet possible de façonner les modélisations cérébrales en utilisant des données structurelles acquises empiriquement, après avoir analysé par IRM le cerveau d’un individu donné par exemple. Autrement dit, la forme du cerveau et la connectivité des neurones d’une personne peuvent être utilisées pour façonner un réseau cérébral qui lui sera propre.
Il est possible d’aller encore un pas plus loin : en utilisant des méthodes d’inférence statistiques, ces données acquises empiriquement peuvent être utilisées pour configurer le modèle obtenu, de façon à créer un véritable avatar virtuel du cerveau du patient.
Cette possibilité ouvre des perspectives en matière de médecine personnalisée : il devient envisageable de planifier virtuellement une intervention chirurgicale, ou d’établir le pronostic d’un large éventail de troubles neurologiques ou de maladies neurodégénératives.
Un cas concret : l’épilepsie résistante aux médicaments
Un exemple concerne le traitement chirurgical de patients atteints d’épilepsie résistante aux médicaments. La personne hospitalisée se voit implanter des électrodes afin de réaliser un électroencéphalogramme enregistrant son activité cérébrale durant les crises. Ces données sont censées permettre d’identifier les zones à l’origine de son épilepsie, qui seront traitées chirurgicalement.
Le problème est qu’il peut parfois s’avérer difficile d’intégrer toutes les données et informations collectées, et d’en tirer des informations claires. La création d’un modèle de réseau cérébral basé sur une IRM du cerveau du malade permettrait de lever cette ambiguïté. Grâce à elle, il deviendrait en effet possible d’intégrer les informations disponibles de façon à mettre en évidence clairement des associations entre certaines données qui seraient difficiles à appréhender à l’œil nu : propagation de l’influx nerveux à travers une zone non monitorée, effet du placement des électrodes, etc.
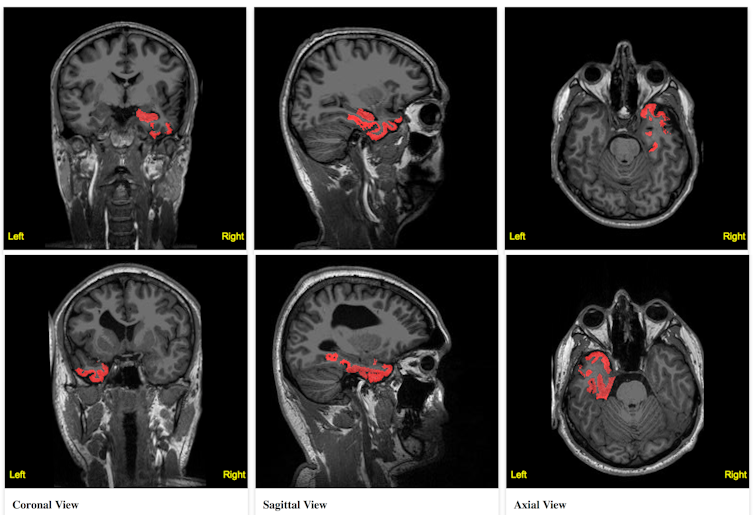
Dans un premier temps, les données IRM seraient utilisées pour définir la forme et la connectivité du cerveau virtuel. On ajouterait ensuite sur ce modèle des électrodes (virtuelles elle aussi), aux mêmes positions que dans la réalité. Grâce à un modèle dynamique capable de simuler la transition entre les états relâchés et épileptiques, on pourrait alors régler finement les paramètres des différentes régions de ce cerveau virtuel : l’objectif serait que la simulation reproduise exactement les enregistrements réalisés sur le patient.
Une telle procédure permettrait d’identifier les régions les plus impliquées dans la survenue des crises, fournissant aux cliniciens qui vont intervenir des données complémentaires pour éclairer leurs décisions.
Cette approche a déjà permis d’obtenir des résultats encourageants durant des études pilotes. Elle est actuellement évaluée dans le cadre d’une étude clinique prospective au sein du projet EPINOV.
Le graal : les modèles multiéchelles
Le but ultime des neurosciences computationnelles est de parvenir à intégrer les données et les connaissances sur le cerveau à différentes échelles spatiales et temporelles, des neurones à l’organe entier, de durées allant de la milliseconde à plusieurs décennies.
Dans le cadre des modélisations du cerveau entier, il s’agit de parvenir à construire un modèle de réseau cérébral performant dans plus d’un scénario. Celui-ci permettrait serait capable par exemple de saisir les caractéristiques de l’état de repos non seulement selon la modalité employée pour l’examen (à la fois lors de l’utilisation non seulement de l’électroencéphalogramme, mais aussi de l’IRM, par exemple) ou lorsque le sujet s’adonne à diverses tâches (ce modèle devrait par exemple être capable de rendre compte correctement à la fois de l’état de repos et de la réponse à une stimulation par exemple).
S’il était dans le même temps possible de fusionner les modèles « top-down » (ceux représentant des réseaux de populations de neurones) et « bottom-up » (ceux simulant des réseaux de neurones individuels) en une seule instance multiéchelle, on obtiendrait le meilleur des deux mondes : ce modèle permettrait de simuler le fonctionnement du cerveau entier tout en permettant d’observer en détail quelques régions spécifiques, représentées de façon réaliste.
Un tel modèle ouvrirait de nouvelles perspectives pour les expérimentations « in silico » (c’est-à-dire des simulations numériques) visant à comprendre les conséquences de changements locaux, documentés par des données empiriques sur la fonction cérébrale globale. Mais la mise au point de modèles multiéchelles se heurte à plusieurs problèmes conceptuels et sociotechniques.
D’importants défis à relever
Si les modèles dits « generative brain network » (modèles cérébraux « génératifs »), basés sur la théorie, ont l’avantage d’être plus interprétables, ils sont moins robustes que les approches dénuées de modèles. Si, lors de la conception, on commet une erreur à propos d’un des composants d’un modèle génératif (si on utilise une population neuronale inappropriée, mise au point pour une autre expérimentation, par exemple), le modèle peut s’avérer incapable de reproduire correctement les données empiriques.
Le problème est que les conséquences n’émergeront que sur le long terme : c’est au moment où le modèle ne fonctionnera plus que l’on réalisera qu’il existe des lacunes dans la compréhension du phénomène étudié. Imaginons par exemple qu’un tel modèle génératif indique qu’une activité donnée ne fonctionne que selon une fréquence dominante unique, dans l’ensemble du cerveau. Si des données empiriques révèlent au contraire l’existence d’une variabilité de cette fréquence dominante dans certaines régions cérébrales, alors le modèle devra être revu.
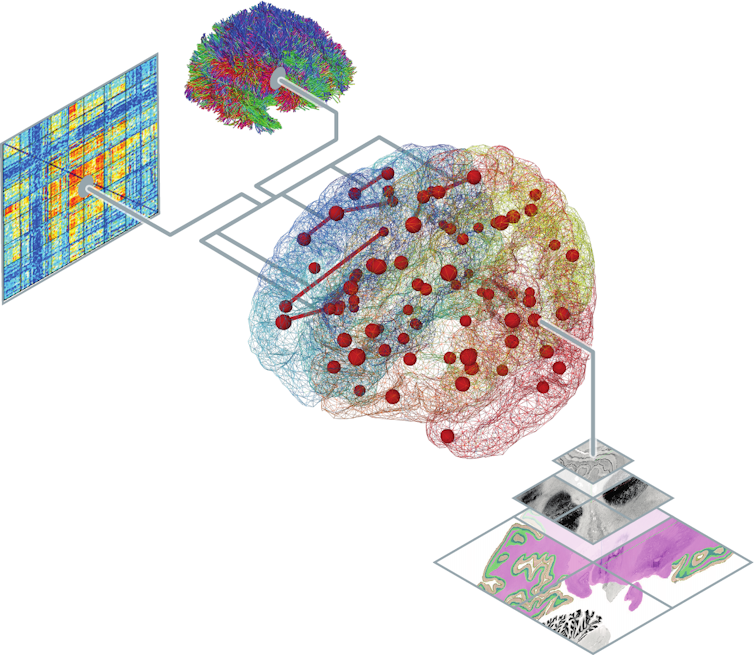
Combler ces lacunes requiert un important effort interdisciplinaire. Il s’agit de recruter des compétences dans un large éventail de domaines, de la physique à la neuroimagerie (pour l’acquisition et le traitement de données) en passant par les mathématiques (pour la formulation et l’analyse de modèles) et de nombreuses branches des neurosciences (cliniques, cognitives, etc.).
Dans le cas des modèles multiéchelles, c’est le couplage entre les différents niveaux de détail qui est compliqué à mettre en place. D’un côté, nous avons des modèles portant sur une seule population de neurones simulée finement, en se basant sur les neurones individuels qui la constituent et les connexions élaborées qu’ils établissent les uns avec les autres, via lesquelles ils communiquent entre eux. De l’autre côté, nous avons des modèles représentant des populations neuronales décrites en termes de variables sommaires, telles que l’activité moyenne des neurones qui les composent et leur connectivité avec les régions voisines, représentée relativement grossièrement.
Pour pouvoir « dézoomer », autrement dit passer de l’échelle inférieure à l’échelle supérieure, il faut pouvoir agréger de grandes quantités détails fins pour les faire correspondre à l’image globale. Pour « zoomer », au contraire, il faut être capable de faire correspondre des statistiques sommaires (par exemple la vitesse moyenne de déclenchement des neurones) avec des données dont le grain est très fin (comme celles correspondant à la génération d’un flux de pics constant). Passer d’un niveau de détail à l’autre est difficile, non seulement quand il s’agit de représenter la connectivité des neurones, mais aussi pour ce qui est de rendre compte des informations véhiculées.
Par ailleurs, la mise en œuvre d’une simulation d’une telle complexité représente du point de vue de la performance logicielle. La mise au point d’infrastructures capables de traiter de telles données implique en effet de relever plusieurs défis : ces systèmes doivent être capables de traiter de grands volumes de données en assurant leur sécurisation, tout en étant conformes aux diverses réglementations légales et éthiques, lesquelles varient d’un pays à l’autre.
On l’a vu, la confrontation des modèles numériques avec les données empiriques est un point important pour s’assurer de leur validité. Cette validation peut être effectuée en construisant des cohortes virtuelles de patients – autrement dit en traitant des groupes composés de nombreux individus. De telles cohortes peuvent être par exemple mises en place pour comprendre comment le cerveau change au fil du vieillissement. Pour être validés, les modèles de cerveau personnalisés doivent s’avérer capables de rendre compte des caractéristiques les plus importantes dudit vieillissement avec un pouvoir prédictif suffisant.
Pour toutes ces raisons, de tels projets ne peuvent être menés qu’au sein de collaborations de grande ampleur, comme celle mise en œuvre pour développer l’infrastructure de recherche EBRAINS pour les neurosciences du Human Brain Project.
L’ensemble de ces efforts est le prix à payer pour parvenir, à terme, à la mise au point de simulations personnalisées, qui nous permettront non seulement de trouver de nouvelles solutions cliniques, mais aussi de mieux comprendre ce qui fait que nous sommes qui nous sommes…![]()
Jan Fousek, Neuroscientifique, Aix-Marseille Université (AMU) et Viktor Jirsa, Directeur de l’Institut de Neurosciences des Systèmes, Aix-Marseille Université (AMU)
Cet article est republié à partir de The Conversation sous licence Creative Commons. Lire l’article original.
Retrouvez d’autres articles scientifiques sur le site dans la catégorie sciences
